import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import matplotlib as mpl
%matplotlib inline
import networkx as nxNetwork Science - UDD
Network Projections
Cristian Candia-Castro Vallejos, Ph.D.
Yessica Herrera-Guzmán, Ph.D.
- [1] Data Science Institute (IDS), Universidad del Desarrollo,Chile
- [2] Northwestern Institute on Complex Systems, Kellogg School of Management, Northwestern Unviersity, USA
- [3] Center for Complex Network Research (CCNR), Northwestern Unviersity, USA
Número de personas que contrajeron COVID
Este es un conjunto de datos generado aleatoriamente para ilustrar la construcción de redes bipartitas a partir de conjuntos de datos tabulares empíricos.
En este conjunto de datos, hay variantes de COVID y los nombres enumerados para cada variante son aquellos que fueron infectados con esa variante.
El objetivo de esta práctica es que te familiarices con este tipo de datos estructurados que no están en forma de lista de aristas, sino que se asemejan a fuentes de datos de investigaciones empíricas. A partir de estos datos, deberías ser capaz de crear una red bipartita, así como la lista de nodos y la lista de aristas para cada una de las proyecciones de la red.
data = pd.read_csv('./Data/COVIDvariants.csv')data.head()| Variant1 | Variant2 | Variant3 | Variant4 | Variant5 | Variant6 | Variant7 | Variant8 | Variant9 | Variant10 | |
|---|---|---|---|---|---|---|---|---|---|---|
| 0 | Charlie | Frank | Quinn | Oliver | Charlie | Helen | Grace | Rachel | Michael | Grace |
| 1 | Sam | David | David | Nina | Nadia | Bob | Ivy | Laura | Tina | Ronald |
| 2 | Nina | Oliver | Charlie | Helen | Paul | Eva | Quinn | Sam | Ivy | Bob |
| 3 | Alice | Quinn | Bob | Quinn | Sam | Jack | Kevin | Michael | Frank | Nina |
| 4 | Kevin | Sam | Rachel | Jack | Kevin | Frank | Eva | Alice | Quinn | Laura |
Convert the dataset to the edgelist of a bipartite network, where one level (col1 = ‘variant’) are the variants and another level (col2 = ‘name’) are the infected individuals.
df = data.melt(var_name='variant', value_name='name').dropna()df| variant | name | |
|---|---|---|
| 0 | Variant1 | Charlie |
| 1 | Variant1 | Sam |
| 2 | Variant1 | Nina |
| 3 | Variant1 | Alice |
| 4 | Variant1 | Kevin |
| ... | ... | ... |
| 395 | Variant10 | Sarah |
| 396 | Variant10 | Paula |
| 397 | Variant10 | Ted |
| 398 | Variant10 | Arthur |
| 399 | Variant10 | Robert |
221 rows × 2 columns
g = nx.Graph()g = nx.from_pandas_edgelist(df, source='variant', target='name')# print(nx.info(g))
print(g)Graph with 69 nodes and 221 edgesg.degree('Ivy')4g.degree('Variant1')41variants = list(df.variant.unique())people = list(df.name.unique())layout = nx.spring_layout(g, k=0.05, iterations=50)plt.figure(figsize=(12, 12))
# Draw VARIANTS
nx.draw_networkx_nodes(g,
layout,
nodelist=variants,
node_size=80, # a LIST of sizes, based on g.degree
node_color='green',
edgecolors='green',
linewidths=1.5)
# Draw EVERYONE
nx.draw_networkx_nodes(g, layout, nodelist=people,
node_color='navy',
edgecolors='navy',
alpha=0.9,
linewidths=0.7,
node_size=20)
# Draw REINFECTED PEOPLE
reinfected_people = [person for person in people if g.degree(person) > 1]
nx.draw_networkx_nodes(g, layout, nodelist=reinfected_people,
node_color='red',
edgecolors='navy',
linewidths=0.7,
node_size=40)
nx.draw_networkx_edges(g, layout, width=0.7, alpha=0.9,
edge_color="limegreen")
# # label people
# node_labels = dict(zip(people, people))
# nx.draw_networkx_labels(g, layout, labels=node_labels,
# font_size=8, font_weight='400')
# # label variants
# node_variants = dict(zip(variants, variants))
# nx.draw_networkx_labels(g, layout, labels=node_variants,
# font_size=12, font_weight='400')
# Turn off the axis because I know you don't want it
plt.axis('off')
plt.title("Network of COVID variants")
# plt.savefig('network_name.pdf', bbox_inches='tight', pad_inches=0)
# Tell matplotlib to show it
plt.show()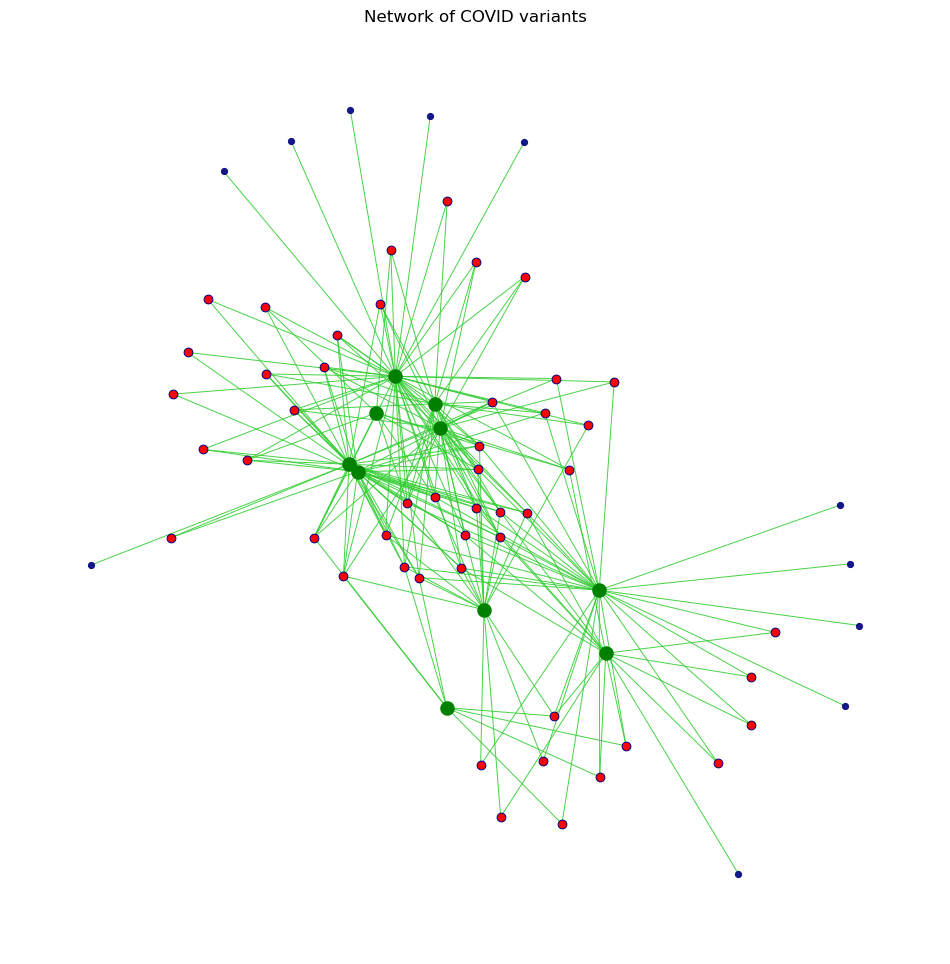
Tarea
Pregunta 1: ¿Cuál es la naturaleza de la relación entre los dos conjuntos de nodos en la red bipartita?
Pregunta 2: ¿Existen medidas específicas para la centralidad e influencia de nodos en redes bipartitas?
Pregunta 3: ¿Cuáles son las redes proyectadas de cada conjunto de nodos? (es decir, redes separadas para cada conjunto)
Ejercicio: Construye la matriz de adyacencia de sus dos proyecciones, en los nodos morados y en los nodos verdes, respectivamente. Vea el ejemplo a continuación. Luego, explique la relevancia de cada proyección.
Project to unipartite: Infected people
wide_df = df.pivot_table(index='variant', columns=df.groupby('variant').cumcount(), values='name', aggfunc='first')wide_df.columns = [f'Name{i+1}' for i in range(wide_df.shape[1])]# Initialize empty lists to store source and target names
source = []
target = []
# Iterate through DataFrame rows
for index, row in wide_df.iterrows():
# Iterate through column names in the current row
for i, name1 in enumerate(row):
for j, name2 in enumerate(row[i+1:]):
# Append source and target names to the lists
source.append(name1)
target.append(name2)
# Create a new DataFrame from the source and target lists
people_df = pd.DataFrame({'Source': source, 'Target': target})people_df = people_df.dropna()new_df = people_df.drop_duplicates()new_df = new_df[new_df['Source'] != new_df['Target']]h = nx.from_pandas_edgelist(new_df, source='Source', target='Target')print(nx.info(h))Graph with 59 nodes and 1265 edgeslayout_h = nx.spring_layout(h, k=0.1, iterations=50)labels = {node: h.nodes[node].get('label', f'Node {node}') for node in h.nodes}plt.figure(figsize=(12, 12))
nx.draw_networkx_nodes(h,
layout_h,
node_size=[(h.degree(v)) ** 2 for v in h.nodes()], # sizes based on h.degree
node_color='lavender',
edgecolors='rebeccapurple',
linewidths=1)
nx.draw_networkx_edges(h, layout_h, width=0.4, alpha=0.7, edge_color="gray")
node_labels = dict(zip(labels, labels))
nx.draw_networkx_labels(h, layout_h, labels=node_labels,
font_size=8)
plt.axis('off')
plt.title("Network of Infected People")
# plt.savefig('network_projection_name.pdf', bbox_inches='tight', pad_inches=0)
plt.show()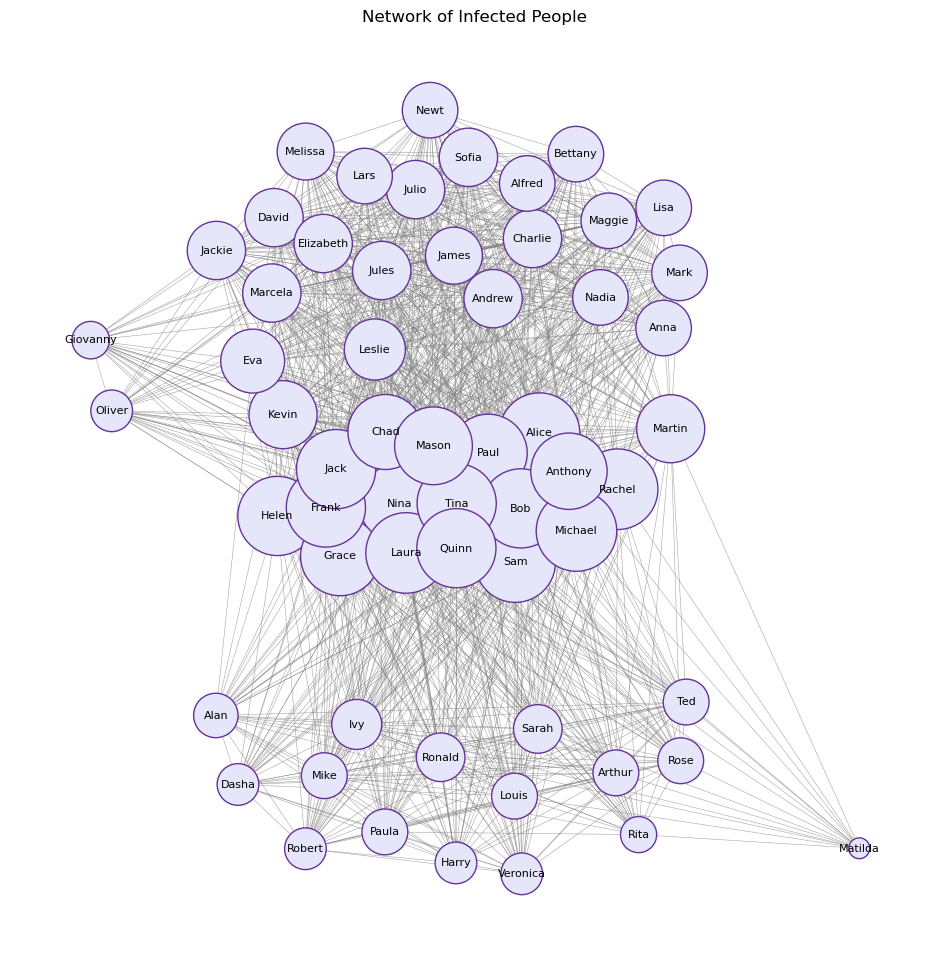
Tarea
Pregunta 1: ¿Cuáles son las redes proyectadas de cada conjunto de nodos? (es decir, redes separadas para cada conjunto)
Pregunta 2: ¿Qué información puedes obtener de cada proyección?
Pregunta 3: Explica la utilidad de las proyecciones de redes con respecto a la visualización y análisis de la red bipartita.
Ejercicio 1: Calcula el grado promedio de cada proyección de la red, luego calcula el grado promedio de los nodos morados y los nodos verdes en la red bipartita. ¿Cuáles son las diferencias en la distribución? Por favor, explica por qué.
Ejercicio 2: Computa las propiedades de estas redes proyectadas (por ejemplo, tamaño, densidad, diámetro, distribución de grados).
Ejercicio 3: Explora visualmente subgrupos o comunidades densamente conectados dentro de las proyecciones. ¿Cómo se relacionan los subgrupos densamente conectados en cada proyección con la red bipartita original?