import numpy as np
import matplotlib.pyplot as plt
import matplotlib as mpl
import networkx as nxNetwork Science - UDD
Básicos en redes
Cristian Candia-Castro Vallejos, Ph.D.
Universidad del Desarrollo (UDD), Chile
Director at the Computational Research in Social Sciences Laboratory (CRiSS-Lab)
Faculty, Data Science Institute, School of Engineering
Faculty, Center for Social Complexity, School of Government
Northwestern University, United States
- External Faculty, Northwestern Institute on Complex Systems (NICO) Kellogg School of Management.
Importando Modulos Requeridos
# !pip install networkx# default settings para plotear Redes
mpl.rc('xtick', labelsize=14, color="#222222")
mpl.rc('ytick', labelsize=14, color="#222222")
mpl.rc('font', **{'family':'sans-serif','sans-serif':['Arial']})
mpl.rc('font', size=16)
mpl.rc('xtick.major', size=6, width=1)
mpl.rc('xtick.minor', size=3, width=1)
mpl.rc('ytick.major', size=6, width=1)
mpl.rc('ytick.minor', size=3, width=1)
mpl.rc('axes', linewidth=1, edgecolor="#222222", labelcolor="#222222")
mpl.rc('text', usetex=False, color="#222222")Tipos básicos de data en NetworkX
NetworkX proporciona las siguientes clases que representan datos relacionados con la red, así como algoritmos de análisis de red que operan en estos objetos:
Graph - Grafos no dirigidos con auto-loops
DiGraph - Grafos dirigidos con auto-loops
MultiGraph - Grafo no-dirigidos con auto-loops y enlaces múltiples
MultiDiGraph - Grafos dirigidos con auto-loops y enlaces múltiples
Comencemos
Crea una red vacía y no-dirigida
G = nx.Graph()G<networkx.classes.graph.Graph at 0x1199a2470>G.nodes()NodeView(())Nodos
Los nodos pueden ser casi cualquier cosa, incluyendo nombres, strings, coordenadas GPS, tú los nombras!
Es posible agregar nodos uno a la vez:
G.add_node(0)
G.add_node("Juan")
# objeto tupla representando, por ejemplo, longitud y latitud
G.add_node((1.2, 3.4))G.nodes()NodeView((0, 'Juan', (1.2, 3.4)))… o muchos a la vez desde un contenedor de python
# [1,2,3] es una lista que contiene 1, 2 y 3
G.add_nodes_from([1, 2, 3,"Carlos"])G.nodes()NodeView((0, 'Juan', (1.2, 3.4), 1, 2, 3, 'Carlos'))Atributos de los Nodos
Los nodos pueden tener atributos arbitrarios asociados a ellos, contenidos en un diccionario string-index
Agragar atributos en el momento en que creamos un nodo usando argumentos:
G.add_node("Jorge", color_ojos='verdes', estatura=175)G.nodes(data=True)NodeDataView({0: {}, 'Juan': {}, (1.2, 3.4): {}, 1: {}, 2: {}, 3: {}, 'Carlos': {}, 'Jorge': {'color_ojos': 'verdes', 'estatura': 175}})Tambien puedes agregar atributos a un nodo existente:
G.add_node("Camila")
G.nodes['Camila']{}G.nodes["Camila"]["color_ojos"] = "rojo"
G.nodes["Camila"]["edad"] = "23"G.nodes["Camila"]{'color_ojos': 'rojo', 'edad': '23'}G.nodes(data=True)NodeDataView({0: {'color_ojos': 'rojo'}, 'Juan': {}, (1.2, 3.4): {}, 1: {}, 2: {}, 3: {}, 'Carlos': {}, 'Jorge': {'color_ojos': 'verdes', 'estatura': 175}, 'Camila': {'color_ojos': 'rojo', 'edad': '23'}})G.add_node("Camila")#, color_ojos='verdes', estatura=175)G.nodes(data=True)NodeDataView({0: {'color_ojos': 'rojo'}, 'Juan': {}, (1.2, 3.4): {}, 1: {}, 2: {}, 3: {}, 'Carlos': {}, 'Jorge': {'color_ojos': 'verdes', 'estatura': 175}, 'Camila': {'color_ojos': 'rojo', 'edad': '23'}})G.node[n] da un diccionario que contiene todos los atributos: pares de valores asociados con el nodo n
print("Los ojos de Jorge son ", G.nodes["Jorge"]["color_ojos"], "y el mide ", G.nodes['Jorge']['estatura'], " centímetros.")
print("Camila tiene los ojos de color", G.nodes["Camila"]["color_ojos"], ".")Los ojos de Jorge son verdes y el mide 175 centímetros.
Camila tiene los ojos de color rojo .Enlaces
Un enlace entre el nodo 1 y el nodo 2 es representado por una tupla (nodo 1, nodo 2).
Ellos pueden ser agregados uno a la vez:
# agrega un enlace entre el nodo 0 y el nodo 1
G.add_edge(0, 1)
G.add_edge('Camila', 100)G.nodes(data=True)NodeDataView({0: {'color_ojos': 'rojo'}, 'Juan': {}, (1.2, 3.4): {}, 1: {}, 2: {}, 3: {}, 'Carlos': {}, 'Jorge': {'color_ojos': 'verdes', 'estatura': 175}, 'Camila': {'color_ojos': 'rojo', 'edad': '23'}, 100: {}})G.edges()EdgeView([(0, 1), ('Camila', 100)])O mucho a la vez desde un contenedor:
edge_list = [ (2, 1), ("Jorge", "Camila"), (3, 4), ("A","B") ]
G.add_edges_from(edge_list)print(G.edges())
# print(G.nodes())[(0, 1), (1, 2), (3, 4), ('Jorge', 'Camila'), ('Camila', 100), ('A', 'B')]Nota: Los nodos será creados automáticamente si ellos no existen aún.
Atributos de los Enlaces
Como para los nodos, los enlaces pueden tener atributos arbitrarios. Un atributo importante y especial (para muchos algoritmos) es el “peso”
La sintaxis para agregar/accesar a los atributos de los enlaces es similar a la de los nodos:
G.add_edge("Jorge", "Claudia", weight=10) # weight en general es un string reservado para algunos algortimos de cerntralidad y comunidades en red.
G.add_edge("Jorge", "Claudia", distancia=20)
G.add_edge("Concepcion", "Santiago")
G.add_edge("Concepcion","Santiago", distancia=510)print(G.edges(data=True))
print('----')
print(G.edges["Concepcion","Santiago"])[(0, 1, {}), (1, 2, {}), (3, 4, {}), ('Jorge', 'Camila', {}), ('Jorge', 'Claudia', {'weight': 10, 'distancia': 20}), ('Camila', 100, {}), ('A', 'B', {}), ('Concepcion', 'Santiago', {'distancia': 510})]
----
{'distancia': 510}G.edges['node1','node2'] es un diccionario que contiene todos los atributos: pares de valores asociados con el enlace del nodo 1 al nodo 2.
Operaciones Básicas
Tamaño de la red
# Número de nodos
print(G.number_of_nodes())
# alternativa
print(len(G))
####################
# Número de enlaces
print(G.number_of_edges())
# alternativa
print(G.size())
########################
# cómo hacer formateo de strings
print("El grafo G tiene {0} nodos y {1} enlaces.".format(len(G), G.size()))16
16
8
8
El grafo G tiene 16 nodos y 8 enlaces.Testeando si nodos y enlaces existen
G.has_node("Jorge")TrueAlternativa pythonica
"Claudia" in GTruePara los enlaces, tu debes usar has_edges (no hay una sintaxisis como para nos lodos edge in G) =(
print(G.has_edge(3, 4))
print(G.has_edge("Jorge", 0))True
FalseEncontrando los vecinos de un nodo
print(list(G.neighbors("Camila")))
print(list(G.neighbors(1)))[100, 'Jorge']
[0, 2]- En un objeto
DiGraph,G.neighbors(node)entrega el nodo sucesor denode, como lo haceG.successors(node)(enlace out) - Predecesores de
nodepueden ser obtenidos conG.predecessors(node)
Iterando sobre los nodos y enlaces
Nodos y enlaces pueden ser iterados con G.nodes() y G.edges() respectivamente.
list(G.nodes(data=True))[(0, {'color_ojos': 'rojo'}),
('Juan', {}),
((1.2, 3.4), {}),
(1, {}),
(2, {}),
(3, {}),
('Carlos', {}),
('Jorge', {'color_ojos': 'verdes', 'estatura': 175}),
('Camila', {'color_ojos': 'rojo', 'edad': '23'}),
(100, {}),
(4, {}),
('A', {}),
('B', {}),
('Claudia', {}),
('Concepcion', {}),
('Santiago', {})]for nodo, atributos in list(G.nodes(data=True)): # data=True incluye atributos de nodo como diccionarios
print("Nodo {0}\t\t\t: {1}".format(nodo, atributos))Nodo 0 : {'color_ojos': 'rojo'}
Nodo Juan : {}
Nodo (1.2, 3.4) : {}
Nodo 1 : {}
Nodo 2 : {}
Nodo 3 : {}
Nodo Carlos : {}
Nodo Jorge : {'color_ojos': 'verdes', 'estatura': 175}
Nodo Camila : {'color_ojos': 'rojo', 'edad': '23'}
Nodo 100 : {}
Nodo 4 : {}
Nodo A : {}
Nodo B : {}
Nodo Claudia : {}
Nodo Concepcion : {}
Nodo Santiago : {}list(G.edges(data=True))[(0, 1, {}),
(1, 2, {}),
(3, 4, {}),
('Jorge', 'Camila', {}),
('Jorge', 'Claudia', {'weight': 10, 'distancia': 20}),
('Camila', 100, {}),
('A', 'B', {}),
('Concepcion', 'Santiago', {'distancia': 510})]for n1, n2, atributo in list(G.edges(data=True)):
print("El primer nodo {0} <----> con el nodo {1}: el atributo es {2}".format(n1, n2, atributo))El primer nodo 0 <----> con el nodo 1: el atributo es {}
El primer nodo 1 <----> con el nodo 2: el atributo es {}
El primer nodo 3 <----> con el nodo 4: el atributo es {}
El primer nodo Jorge <----> con el nodo Camila: el atributo es {}
El primer nodo Jorge <----> con el nodo Claudia: el atributo es {'weight': 10, 'distancia': 20}
El primer nodo Camila <----> con el nodo 100: el atributo es {}
El primer nodo A <----> con el nodo B: el atributo es {}
El primer nodo Concepcion <----> con el nodo Santiago: el atributo es {'distancia': 510}Calculando grados
# one node
print(G.degree("Jorge")) # retorna un entero
# todos los nodos (retorna un diccionario con pares nodo : grado para todos los nodos)
print(G.degree())
# solo la secuencia de grados
print(list(dict(G.degree()).values()))2
[(0, 1), ('Juan', 0), ((1.2, 3.4), 0), (1, 2), (2, 1), (3, 1), ('Carlos', 0), ('Jorge', 2), ('Camila', 2), (100, 1), (4, 1), ('A', 1), ('B', 1), ('Claudia', 1), ('Concepcion', 1), ('Santiago', 1)]
[1, 0, 0, 2, 1, 1, 0, 2, 2, 1, 1, 1, 1, 1, 1, 1]G.edges("Jorge")EdgeDataView([('Jorge', 'Camila'), ('Jorge', 'Claudia')])Como sabes, en un grafo dirigido (de clase DiGraph) hay dos tipos de grados. Las cosas funcionan tal cual como tu lo esperas: * G.in_degree(node) * G.out_degree(node) # igual que G.degree()
Otras operaciones
subgraph(G, nbunch)orG.subgraph(nbunch)
subgrafo de G inducido por los nodos en nbunchreverse(G)
DiGraph con enlaces al revésunion(G1, G2)
unión de grafosdisjoint_union(G1, G2)
Igual, pero con trata a los nodos de G1 y G2 como distintosintersection(G1, G2)
grafo con solo los enlaces en común entre G1 y G2difference(G1, G2)
grafo con solo los enlaces G1 que no estan en G2copy(G)orG.copy()
copia de Gcomplement(G)orG.complement()
El complemento del grafo Gconvert_to_undirected(G)orG.to_undirected()
versión no-dirigida de G (un grafo o multigrafo)convert_to_directed(G)orG.to_directed()
versión dirigida de G (un digrafo o multigrafo)adjacency_matrix(G)
Matriz de adjacencia A del grafo G (en formato de matriz sparse; para obtener la matriz completa (con las entradas iguales a cero, usa A.toarray() )
Graph I/O
Habitualmente, tú no construiras una red desde cero, un nodo link a la vez. Por el contrario, tú querras leer un la red desde un archivo. NetworkX puede entender los siguientes formatos de grafos:
- edge lists
- adjacency lists
- GML
- GEXF
- Python ‘pickle’
- GraphML
- Pajek
- LEDA
- YAML
Comencemos: Leyendo un edge list
Coloca el archivo test.txt en tu directorio de trabajo para IPython. Si no sabes el directorio de trabajo puedes accederlo tecleando %pwd
en cualquier celda
%pwd'/Users/crcandia/Dropbox (Personal)/Cursos_UDD/sitio_networkScience/hands-on/HandsOn_Basics'Lee en el archivo con las siguientes opciones: * Lineas que comienzan con # son tratadas como comentarios e ignoradas * Usa un objeto Graph para almacenar la data (si la red es no-dirigida)
* La data es separada por espacios blancos (’ ’) * Los nodos deberían ser tratados como enteros (int) * El encoding del texto of the text file containing the edge list is utf-8
# lee una edge list desde el archivo'test.txt'
G = nx.read_edgelist('./Data/test.txt',
comments='#',
create_using=nx.Graph(),
delimiter=' ',
# nodetype=int,
encoding='utf-8')Formatos permitidos
- Pares de nodos sin data
1 2 - Pares de nodos con un diccionario de python
1 2 {weight:7, color:"verde"}
Análisis Básico
Un gran número de análisis básicos pueden ser hechos en una linea usando NetworkX (en general usamos nx como alias) + numpy (en general usamos np como alias) o funciones de python como min, max, etc.
G.degree()DegreeView({'0': 5, '482': 2, '162': 5, '499': 5, '362': 4, '389': 3, '1': 2, '356': 2, '244': 1, '2': 2, '347': 2, '227': 3, '3': 5, '184': 2, '278': 2, '340': 2, '142': 3, '30': 3, '4': 1, '124': 3, '5': 5, '493': 8, '65': 3, '319': 6, '293': 7, '193': 6, '6': 3, '80': 1, '273': 3, '491': 3, '7': 1, '127': 1, '8': 2, '49': 4, '405': 3, '9': 2, '219': 4, '85': 5, '10': 1, '395': 3, '11': 2, '192': 7, '182': 6, '12': 3, '352': 1, '439': 2, '199': 5, '13': 3, '235': 3, '341': 1, '14': 1, '215': 5, '16': 3, '388': 5, '77': 4, '47': 1, '17': 2, '435': 2, '167': 3, '19': 1, '286': 1, '22': 2, '164': 4, '140': 1, '23': 2, '196': 5, '94': 1, '24': 4, '402': 2, '411': 3, '173': 3, '326': 3, '26': 1, '453': 4, '29': 1, '97': 1, '320': 3, '31': 3, '360': 4, '497': 3, '243': 1, '33': 1, '470': 2, '34': 2, '400': 1, '134': 4, '35': 1, '259': 5, '36': 2, '111': 6, '37': 3, '496': 2, '76': 6, '79': 3, '38': 1, '40': 1, '88': 5, '41': 4, '200': 4, '73': 4, '92': 2, '481': 3, '42': 3, '445': 2, '486': 3, '44': 3, '190': 2, '119': 3, '45': 3, '186': 2, '163': 3, '198': 2, '46': 3, '252': 3, '48': 1, '176': 3, '129': 2, '250': 3, '50': 3, '336': 3, '122': 3, '51': 3, '224': 1, '434': 2, '299': 1, '52': 1, '342': 4, '53': 1, '498': 1, '54': 2, '474': 5, '55': 2, '382': 3, '334': 5, '56': 3, '339': 1, '123': 3, '438': 6, '57': 2, '177': 2, '451': 1, '58': 1, '59': 1, '120': 2, '60': 2, '385': 2, '260': 3, '61': 3, '83': 3, '133': 4, '454': 3, '62': 2, '242': 2, '255': 4, '63': 3, '432': 2, '107': 2, '64': 3, '328': 3, '221': 3, '67': 1, '386': 3, '68': 5, '369': 2, '178': 2, '323': 2, '141': 2, '69': 3, '401': 2, '70': 1, '99': 4, '428': 1, '74': 4, '276': 2, '154': 2, '469': 3, '75': 2, '329': 5, '132': 2, '214': 2, '442': 1, '158': 3, '370': 2, '407': 3, '383': 2, '78': 3, '137': 3, '282': 2, '292': 3, '81': 1, '82': 2, '165': 7, '139': 3, '477': 2, '272': 4, '121': 2, '86': 1, '480': 4, '253': 5, '90': 3, '194': 4, '101': 3, '91': 1, '93': 1, '96': 1, '268': 4, '98': 2, '426': 2, '335': 3, '289': 3, '467': 3, '207': 4, '100': 2, '468': 4, '267': 5, '102': 1, '384': 3, '103': 4, '353': 3, '463': 4, '104': 1, '105': 1, '422': 4, '108': 1, '110': 1, '213': 2, '279': 1, '136': 3, '436': 2, '112': 3, '440': 4, '456': 3, '324': 1, '114': 1, '327': 3, '115': 1, '344': 2, '116': 3, '322': 2, '187': 3, '310': 3, '117': 4, '201': 2, '131': 3, '265': 5, '241': 2, '118': 1, '203': 2, '387': 4, '297': 4, '125': 2, '240': 4, '126': 2, '285': 5, '238': 2, '128': 4, '378': 2, '183': 2, '130': 2, '325': 4, '419': 3, '228': 2, '304': 1, '484': 2, '135': 1, '364': 2, '359': 2, '138': 2, '220': 2, '150': 2, '143': 2, '144': 1, '145': 3, '475': 2, '146': 1, '147': 2, '148': 2, '294': 1, '191': 5, '149': 1, '461': 2, '233': 1, '151': 2, '354': 1, '152': 5, '374': 6, '447': 2, '155': 4, '248': 2, '156': 1, '157': 2, '209': 2, '410': 2, '222': 2, '159': 2, '358': 4, '160': 2, '249': 4, '398': 1, '466': 2, '460': 1, '269': 3, '212': 1, '427': 3, '431': 4, '168': 6, '166': 2, '379': 2, '179': 1, '303': 2, '495': 2, '169': 3, '441': 2, '180': 1, '170': 3, '262': 1, '171': 2, '473': 1, '174': 1, '175': 1, '489': 2, '478': 1, '181': 3, '472': 2, '450': 2, '376': 3, '185': 2, '487': 2, '311': 1, '403': 2, '188': 3, '488': 2, '307': 1, '189': 1, '291': 2, '195': 2, '424': 1, '397': 3, '197': 2, '371': 2, '218': 1, '202': 2, '337': 3, '261': 1, '204': 1, '205': 2, '394': 2, '391': 2, '208': 2, '399': 2, '210': 2, '357': 3, '211': 4, '225': 2, '301': 4, '415': 2, '257': 2, '277': 3, '216': 2, '423': 4, '247': 3, '217': 2, '236': 2, '302': 2, '275': 1, '223': 2, '380': 3, '393': 3, '229': 1, '464': 2, '230': 2, '231': 2, '290': 2, '375': 1, '237': 3, '274': 1, '239': 1, '355': 2, '437': 2, '409': 4, '338': 2, '331': 1, '313': 3, '251': 1, '396': 5, '430': 2, '306': 2, '254': 2, '361': 3, '256': 1, '263': 1, '264': 1, '312': 1, '266': 2, '408': 2, '309': 2, '270': 3, '417': 4, '443': 2, '366': 1, '271': 2, '406': 4, '295': 2, '429': 1, '280': 1, '281': 2, '283': 1, '288': 2, '284': 1, '455': 1, '287': 2, '452': 2, '485': 3, '444': 1, '315': 1, '462': 2, '316': 1, '318': 1, '317': 1, '420': 2, '365': 3, '330': 1, '332': 2, '433': 2, '348': 1, '343': 1, '350': 1, '346': 1, '349': 3, '351': 2, '363': 1, '368': 2, '421': 2, '390': 1, '377': 1, '490': 3, '381': 1, '458': 1, '494': 3, '404': 2, '418': 1, '457': 1, '465': 1})N = len(G)
L = G.size()
degrees = list(dict(G.degree()).values())
kmin = min(degrees)
kmax = max(degrees)# degreesprint("Número de nodos: ", N)
print("Número de enlaces: ", L)
print('-------')
print("Grado promedio: ", 2*L/N) #Formula vista en clases (qué sucedía con las redes reales?)
print("Grado promedio (alternativa de calculo)", np.mean(degrees))
print('-------')
print("Grado mínimo: ", kmin)
print("Grado máximo: ", kmax)Número de nodos: 443
Número de enlaces: 540
-------
Grado promedio: 2.4379232505643342
Grado promedio (alternativa de calculo) 2.4379232505643342
-------
Grado mínimo: 1
Grado máximo: 8Ploteando la red
- NetworkX puede dibujar redes usando un gran número de algoritmos de “layout” (diseño)
- Los resultados no son tan bonitos como en Gephi, pero NetworkX es mejor para una visualización rápida y no muy pulcra y entrega un control fino sobre el diseño.
G.number_of_edges()540# Usando el algoritmo force-based o "spring"
fig = plt.figure(figsize=(8,8))
nx.draw_spring(G, node_size=40)
# usando el algoritmo circular
fig = plt.figure(figsize=(8,8))
nx.draw_circular(G, node_size=20)
Ploteando la distribución de grados
Vamos a plotearla en escala logaritmica primero (IMPORTANTE!!!!!)
numpy puede ser usado para hacer bines logaritmicamente espaciados entre el grado mínimo y el grado máximo.
print(kmax)
print(kmin)
print(degrees)8
1
[5, 2, 5, 5, 4, 3, 2, 2, 1, 2, 2, 3, 5, 2, 2, 2, 3, 3, 1, 3, 5, 8, 3, 6, 7, 6, 3, 1, 3, 3, 1, 1, 2, 4, 3, 2, 4, 5, 1, 3, 2, 7, 6, 3, 1, 2, 5, 3, 3, 1, 1, 5, 3, 5, 4, 1, 2, 2, 3, 1, 1, 2, 4, 1, 2, 5, 1, 4, 2, 3, 3, 3, 1, 4, 1, 1, 3, 3, 4, 3, 1, 1, 2, 2, 1, 4, 1, 5, 2, 6, 3, 2, 6, 3, 1, 1, 5, 4, 4, 4, 2, 3, 3, 2, 3, 3, 2, 3, 3, 2, 3, 2, 3, 3, 1, 3, 2, 3, 3, 3, 3, 3, 1, 2, 1, 1, 4, 1, 1, 2, 5, 2, 3, 5, 3, 1, 3, 6, 2, 2, 1, 1, 1, 2, 2, 2, 3, 3, 3, 4, 3, 2, 2, 4, 3, 2, 2, 3, 3, 3, 1, 3, 5, 2, 2, 2, 2, 3, 2, 1, 4, 1, 4, 2, 2, 3, 2, 5, 2, 2, 1, 3, 2, 3, 2, 3, 3, 2, 3, 1, 2, 7, 3, 2, 4, 2, 1, 4, 5, 3, 4, 3, 1, 1, 1, 4, 2, 2, 3, 3, 3, 4, 2, 4, 5, 1, 3, 4, 3, 4, 1, 1, 4, 1, 1, 2, 1, 3, 2, 3, 4, 3, 1, 1, 3, 1, 2, 3, 2, 3, 3, 4, 2, 3, 5, 2, 1, 2, 4, 4, 2, 4, 2, 5, 2, 4, 2, 2, 2, 4, 3, 2, 1, 2, 1, 2, 2, 2, 2, 2, 2, 1, 3, 2, 1, 2, 2, 1, 5, 1, 2, 1, 2, 1, 5, 6, 2, 4, 2, 1, 2, 2, 2, 2, 2, 4, 2, 4, 1, 2, 1, 3, 1, 3, 4, 6, 2, 2, 1, 2, 2, 3, 2, 1, 3, 1, 2, 1, 1, 1, 2, 1, 3, 2, 2, 3, 2, 2, 1, 2, 3, 2, 1, 1, 2, 2, 1, 3, 2, 2, 1, 2, 3, 1, 1, 2, 2, 2, 2, 2, 2, 3, 4, 2, 4, 2, 2, 3, 2, 4, 3, 2, 2, 2, 1, 2, 3, 3, 1, 2, 2, 2, 2, 1, 3, 1, 1, 2, 2, 4, 2, 1, 3, 1, 5, 2, 2, 2, 3, 1, 1, 1, 1, 2, 2, 2, 3, 4, 2, 1, 2, 4, 2, 1, 1, 2, 1, 2, 1, 1, 2, 2, 3, 1, 1, 2, 1, 1, 1, 2, 3, 1, 2, 2, 1, 1, 1, 1, 3, 2, 1, 2, 2, 1, 1, 3, 1, 1, 3, 2, 1, 1, 1]# Generamos 20 bins espaciados logaritmicamente entre kmin y kmax
bin_edges = np.logspace(np.log10(kmin), np.log10(kmax), num=10)#bins en escala lineal con espaciado logaritmico
# histograma de la data para esos bines
density, _ = np.histogram(degrees, bins=bin_edges, density=True)bin_edgesarray([1. , 1.25992105, 1.58740105, 2. , 2.5198421 ,
3.1748021 , 4. , 5.0396842 , 6.34960421, 8. ])# np.histogram(degrees, bins=bin_edges, density=True)np.log10(bin_edges)array([0. , 0.04753105, 0.0950621 , 0.14259316, 0.19012421,
0.23765526, 0.28518631, 0.33271736, 0.38024842, 0.42777947,
0.47531052, 0.52284157, 0.57037262, 0.61790368, 0.66543473,
0.71296578, 0.76049683, 0.80802788, 0.85555894, 0.90308999])Ahora lo graficamos!
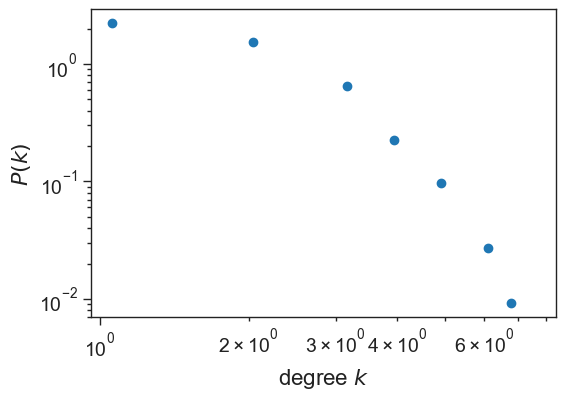
fig = plt.figure(figsize=(6,4))
# "x" debe ser el punto medio (en escala LOG) de cada bin
log_be = np.log10(bin_edges)#debemos hacer el logaritmo para hacer la suma en escala logaritmica
x = 10**((log_be[1:] + log_be[:-1])/2)# calculamos el punto medio (X_i+X_(i+1))/2 y volvemos a la escala anterior.
plt.loglog(x, density, marker='o', linestyle='none')#notacion ejes en logaritmo
plt.xlabel(r"degree $k$", fontsize=16)
plt.ylabel(r"$P(k)$", fontsize=16)
# # remuevo los limites derecho y superior
# ax = plt.gca()
# ax.spines['right'].set_visible(False)
# ax.spines['top'].set_visible(False)
# ax.yaxis.set_ticks_position('left')
# ax.xaxis.set_ticks_position('bottom')
# Muestra la gráfica
plt.show()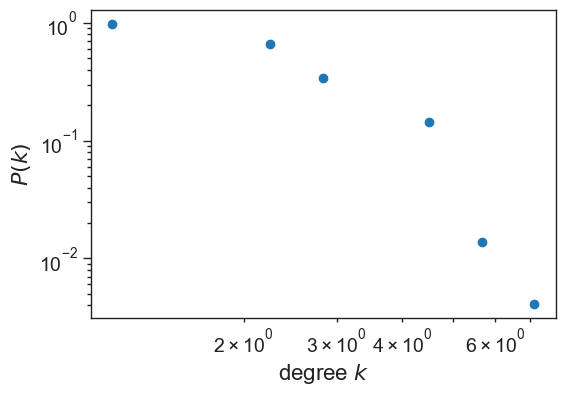
# Chequeamos el espaciado
a=bin_edges[1:] - bin_edges[:-1]
# plt.semilogy(a, color='blue', lw=2)#si el una curva de crecimiento logaritmico, deberia ser una linea recta en un semilog plot
plt.plot(a, color='blue', lw=2)
plt.show()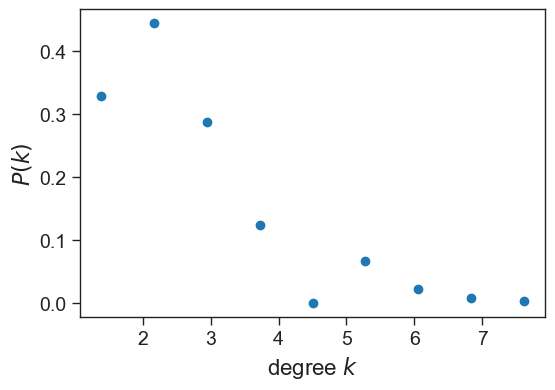
Esto claramente no es una red con algo así como una distribución de grado de ley de potencia o de cola pesada También vamos a graficarla en escala lineal.
El comando linspace de numpy se usa para obtener números linealmente espaciados entre dos extremos.
# Entrega 20 bins linealmente espaceados entre kmin y kmax
bin_edges = np.linspace(kmin, kmax, num=10)
# histograma de la data en estos bines
density, _ = np.histogram(degrees, bins=bin_edges, density=True)Ahora lo graficamos!
fig = plt.figure(figsize=(6,4))
# "x" debería ser el punto medio de cada bin
x = ((bin_edges[1:] + bin_edges[:-1])/2)
plt.plot(x, density, marker='o', linestyle='none')
plt.xlabel(r"degree $k$", fontsize=16)
plt.ylabel(r"$P(k)$", fontsize=16)
# Muestra la gráfica
plt.show()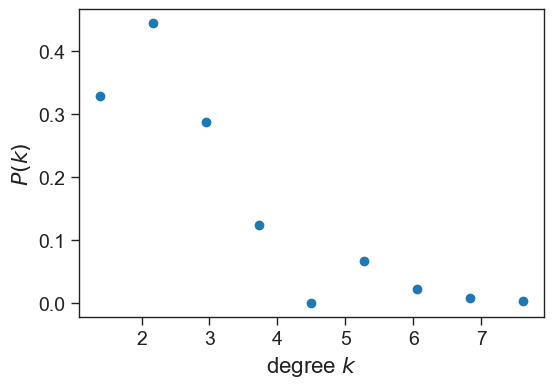
# Chequeamos el espaciado
(bin_edges[1:] - bin_edges[:-1])array([0.25992105, 0.32748 , 0.41259895, 0.5198421 , 0.65496 ,
0.8251979 , 1.0396842 , 1.30992001, 1.65039579])Tarea 1
De manera individual. Tienen una semana para entregar esta tarea. Para ambos datasets example_1.txt y example_2.txt. Cada uno deberá cargar el edge list que corresponde y hacer lo siguiente:
Pregunta 1: Calcula todas las medidas básicas que se muestran arriba. Comente ¿Qué se puede sospechar acerca de la distribución de grados de la red solo en función de la media y los grados extremos?
Pregunta 2: Graficar la distribución de grado en escala log-log. También graficarlo en escala lineal. Comente cómo encaja esto con el análisis de la pregunta 1.
Pregunta 3: Dibuje la red utilizando los dos algoritmos de diseño que se muestran arriba. ¿Cómo se refljan en la apariencia de la red los hallazgos de las preguntas 1 y 2?
Nota: Recuerden que SU interpretación de los resultados es lo más importante.
# Generamos 10 bins espaciados logaritmicamente entre kmin y kmax
bin_edges = np.logspace(np.log10(kmin), np.log10(kmax), num=20)
# histograma de la data para esos bines
density, _ = np.histogram(degrees, bins=bin_edges, density=True)
fig = plt.figure(figsize=(6,4))
# "x" debe ser el punto medio (en escala LOG) de cada bin
log_be = np.log10(bin_edges)
x = 10**((log_be[1:] + log_be[:-1])/2)
plt.loglog(x, density, marker='o', linestyle='none')
plt.xlabel(r"degree $k$", fontsize=16)
plt.ylabel(r"$P(k)$", fontsize=16)
# # remuevo los limites derecho y superior
# ax = plt.gca()
# ax.spines['right'].set_visible(False)
# ax.spines['top'].set_visible(False)
# ax.yaxis.set_ticks_position('left')
# ax.xaxis.set_ticks_position('bottom')
# Muestra la gráfica
plt.show()